Die Auswertungen basieren auf dem Datensatz "SARS-CoV-2-Sequenzdaten aus Deutschland" des Robert-Koch-Instituts. Zur Aktualität dieser Daten siehe auch den Abschnitt "Aktualität" weiter unten.
Dieses Repository ist ein Fork des gleichnamigen Repositories von @corneliusroemer. Für weitere Informationen und eine umfrangreichere README siehe dort.
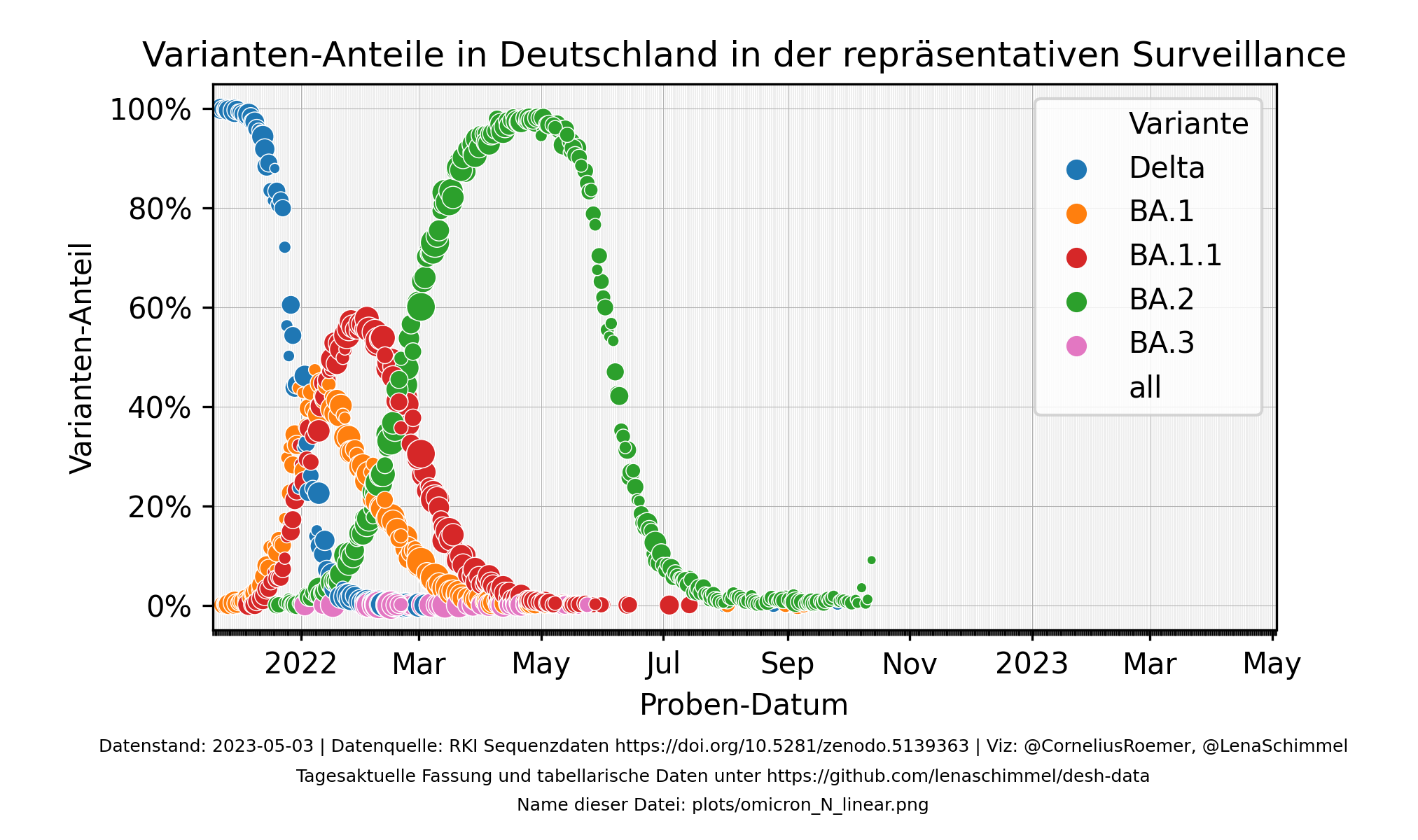
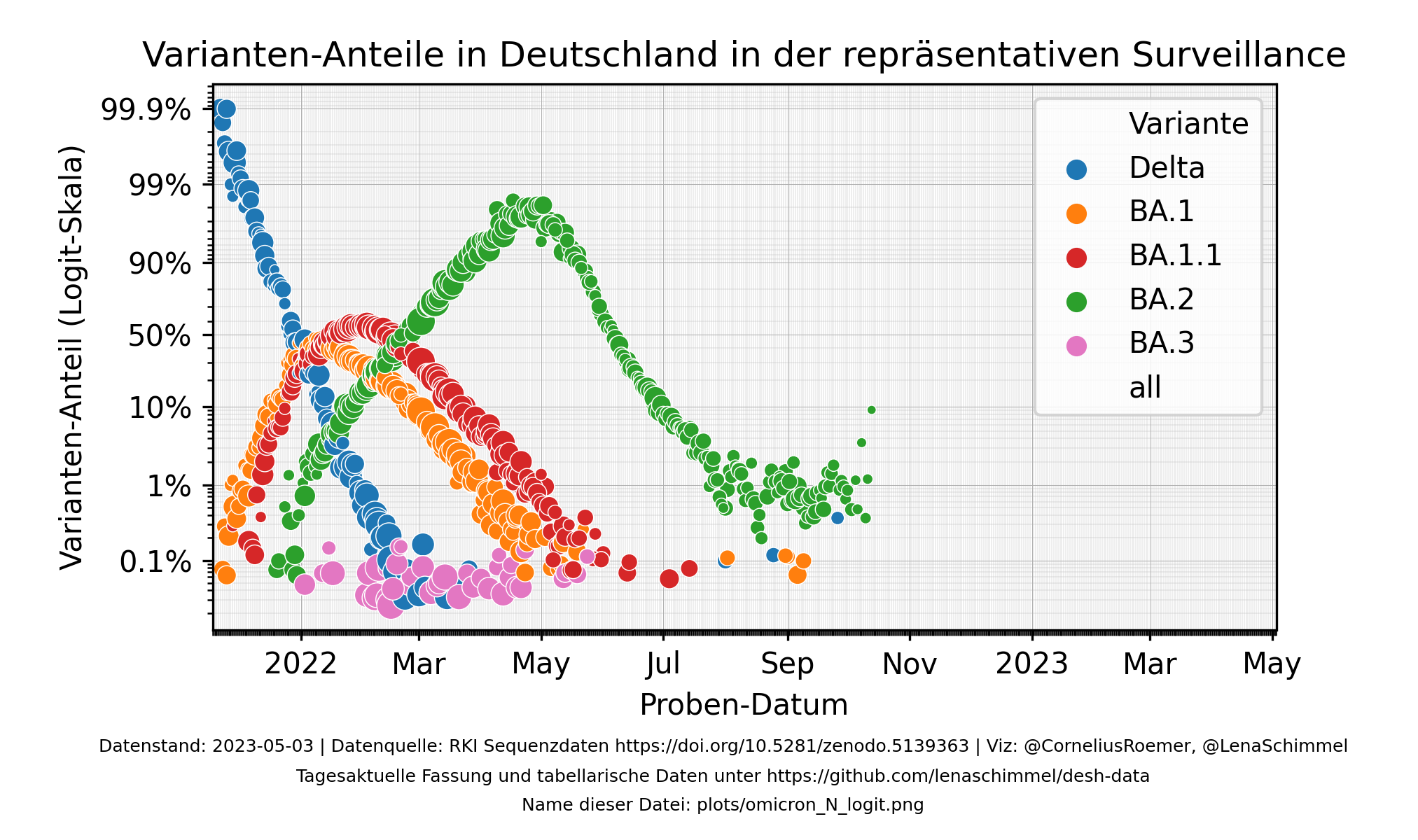
Die unten stehenden Grafiken basieren auf der "Repräsentativen Surveillance" in Deutschland, d.h. aus allen PCR-positiven Proben eines Tages werden zufällig bis zu 5% ausgewählt und Sequenziert. Durch die Sequenzierung kann die Variante genau bestimmt werden.
Die beiden folgenden Grafiken unterscheiden sich nur in der Skalierung der Y-Achse. Die lineare Skala ist einfacher zu lesen, für die Extrapolation des Verlaufs in die Zukunft ist eine Logit-Skala oft praktischer.
Neuerdings werden auch Proben mit der Beschriftung "Unklar" angezeigt. Dies sind Sequenzen, die nicht mit absoluter Sicherheit einer bestimmten Variante zugeordnet werden konnten. Diese hat es immer schon gegeben, aber bisher wurden sie nicht mit in den Grafiken aufgeführt. So gut wie alle dieser Proben gehören nach wie vor zu einer Omicron-Variante (Stand 5.3.22), jedoch ohne absolute Sicherheit, welche genau.
Hinweis: Alle Grafiken auf dieser Seite können durch einen Klick vergrößert werden.
| Linear | Logit |
|---|---|
 |
 |
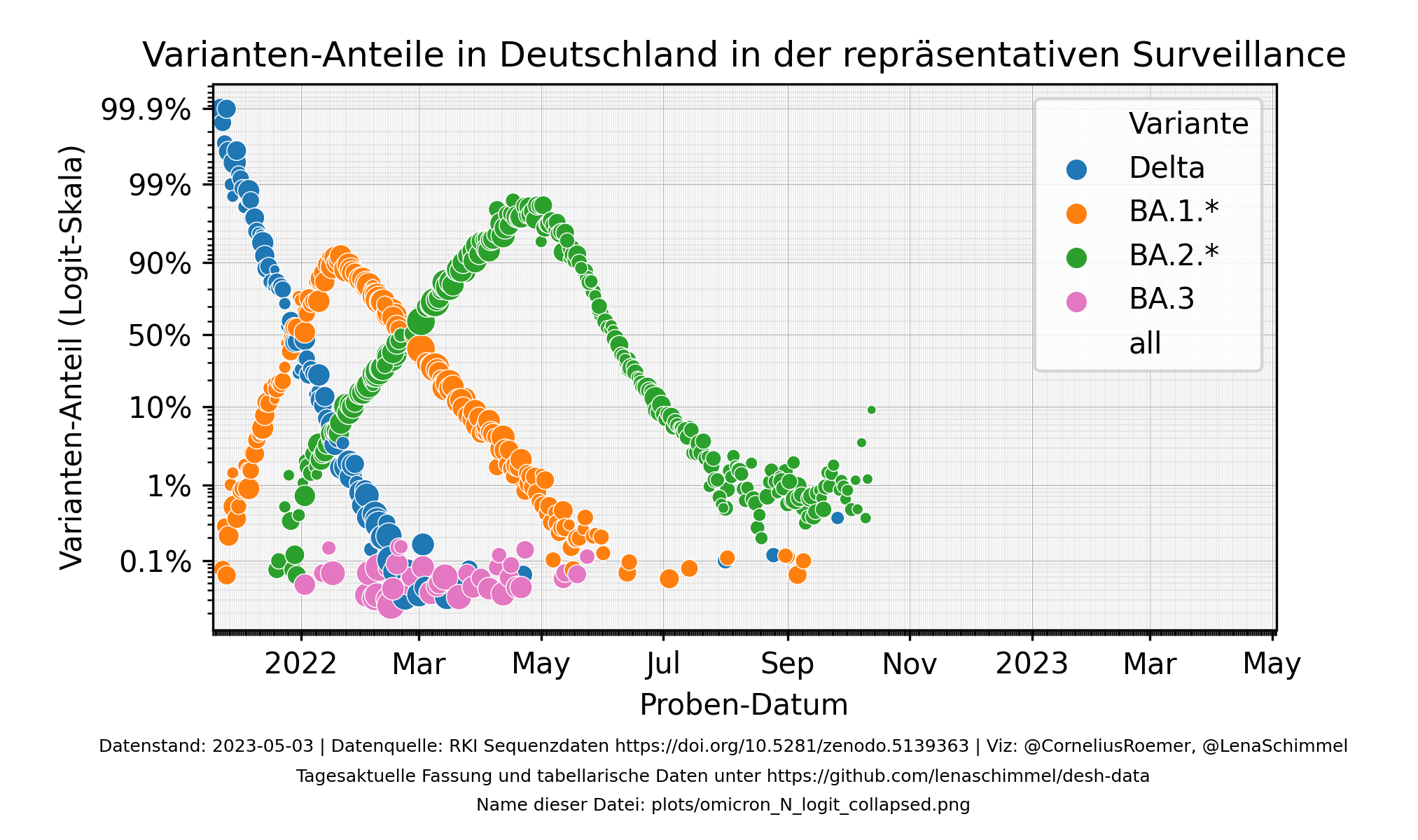
In den folgenden Darstellungen wurden ähnliche Varianten (derzeit nur BA.1 und BA.1.1) zusammen gefasst:
| Linear | Logit |
|---|---|
 |
 |
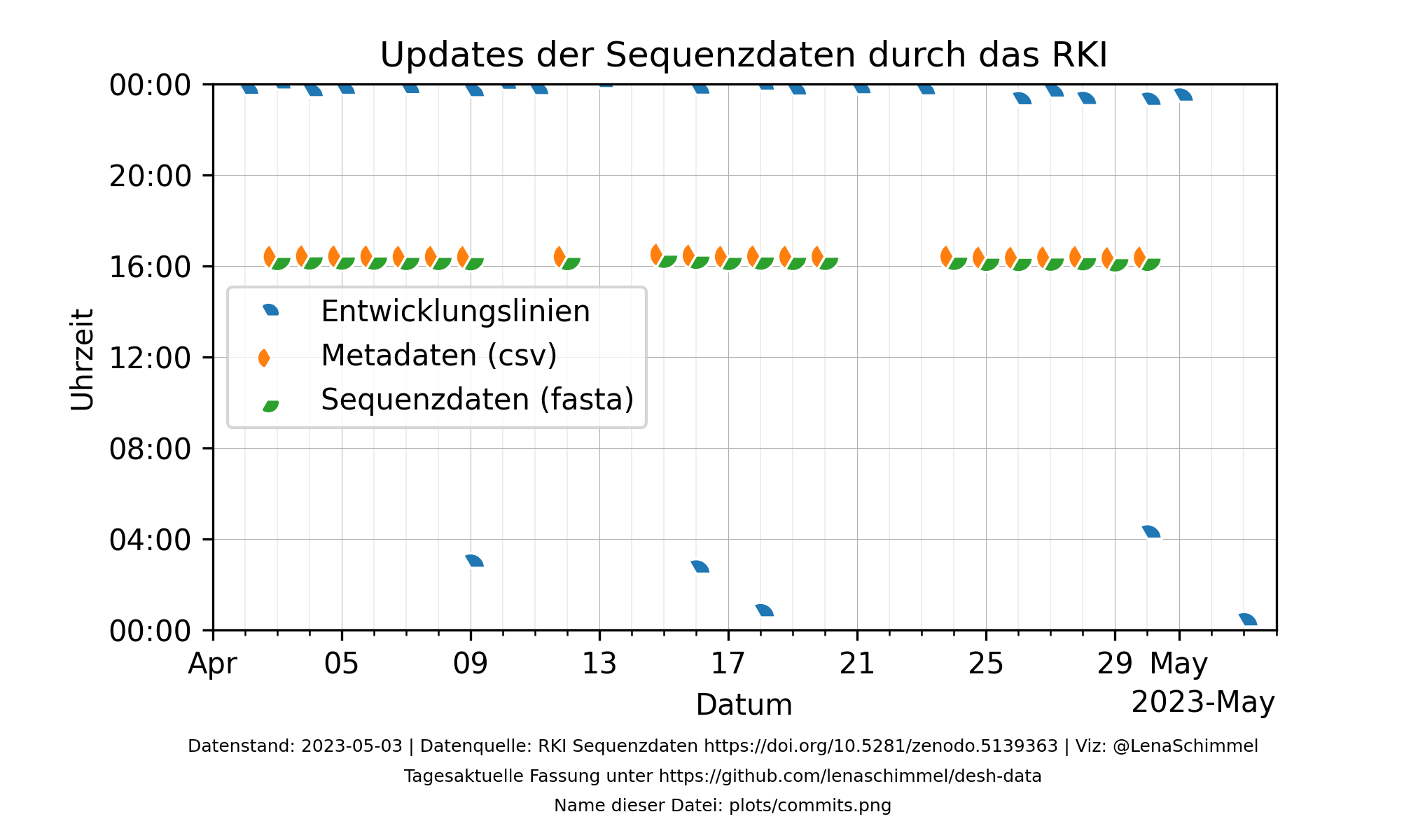
Die Grafiken werden alle zwei Stunden neu generiert, aber ihre Inhalte sind natürlich nur so aktuell wie die zu Grunde liegenden Daten des RKI. Diese werden etwa einmal täglich aktualisiert. Wann genau dies im letzten Monat passiert ist, zeigt die linke Grafik.
Alle Proben werden in den Grafiken nach dem Zeitpunkt der Entnahme dargestellt. Bis zur Sequenzierung vergeht unterschiedlich viel Zeit, so dass die Variantenanteile der letzten paar Tage eine kleinere und somit weniger repräsentative Datenbasis ergibt. Der Verzug ist in der rechten Grafik dargestellt.
| Updates des RKI | Verarbeitungsverzug |
|---|---|
 |
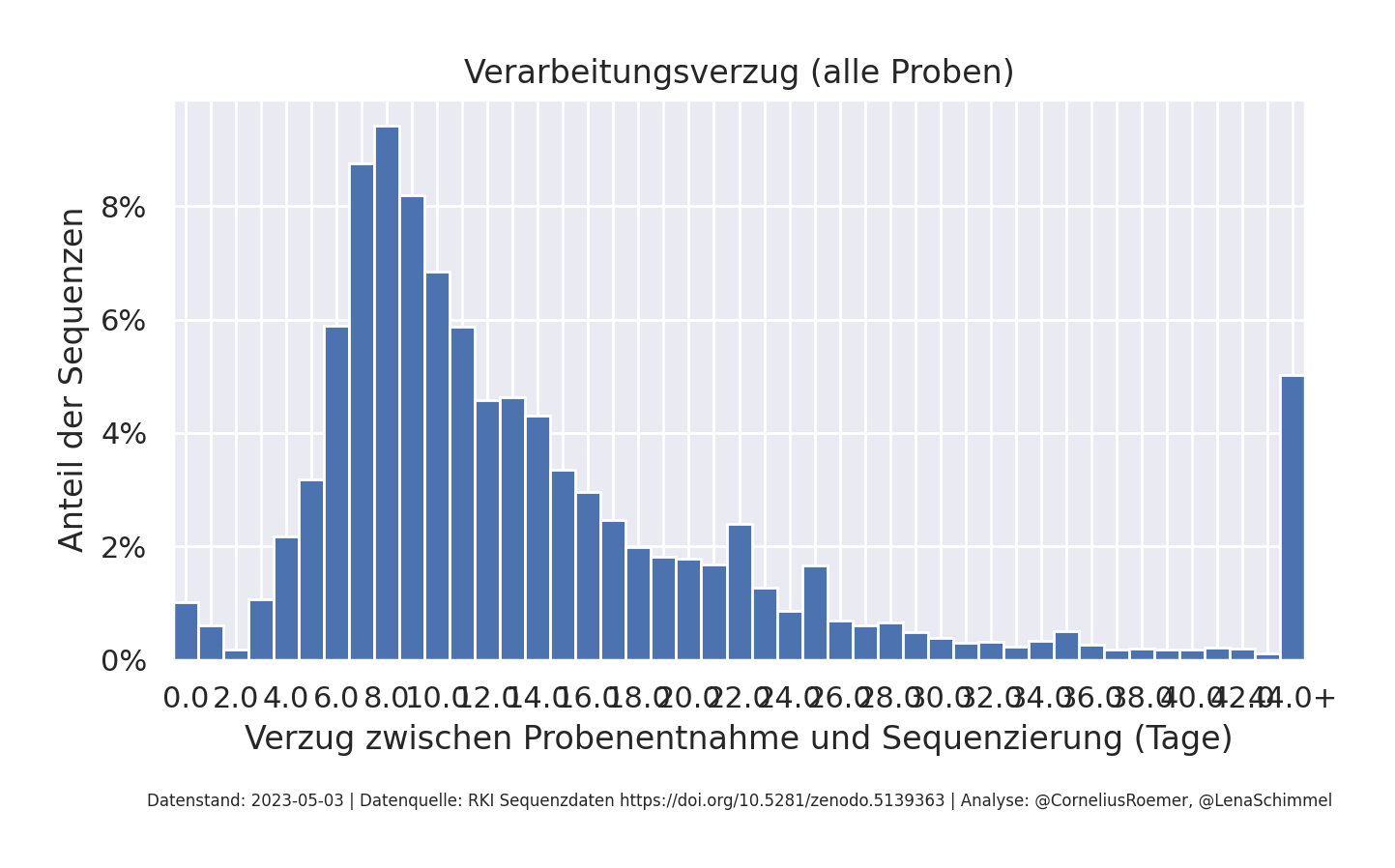 |
Genau genommen sind in den Grafiken und Tabellen nur Sequenzen enthalten, die sowohl in den Entwicklungslinien als auch in den Metadaten vorliegen. Die ältere dieser beiden Dateien gibt somit den tatäschlichen Datenstand dieser Auswertungen vor.
Im Verzeichnis plots befinden sich die die oben gezeigten Grafiken sowie viele weitere Variationen davon. Ein Dateiname wie omicron_N_linear_collapsed.png besteht aus den folgenden Teilen:
omicron_{grund}_{skala}_{collapsed?}.png
| Kürzel | Beschreibung |
|---|---|
| X | (dem sequenzierenden Labor) unbekannt |
| N | Nein (z. B. zufällige Auswahl einer in der PCR positiven Probe zur Sequenzierung) |
| Y | Ja, aber die Art der Mutation bzw. Variante ist (dem sequenzierenden Labor) jedoch unbekannt |
| A | Ja, es besteht aus der vorherigen Diagnostik Verdacht auf die Mutation/Variante [spezifizieren] |
| NX | Zusammenfassung aller Problen mit den Gründen "N" und "X" |
| all | Zusammenfassung aller Proben, unabhängig vom Grund |
| Skala | Beschreibung |
|---|---|
| linear | Gewöhnliche Skala von 0% bis 100% |
| logit | Die Hälten oberhalb und unterhalb von 50% verhalten sich in etwa wie eine logarithmische Skala |
Wenn der Name collapsed enthält, sind ähnliche Varianten zusammen gefasst. Ansonsten werden sie einzeln angegeben.
Im Verzeichnis data/table befinden sich die Daten der Grafiken als CSV-Tabellen. Sie haben stets den selben Datenstand wie die Grafiken. Das Schema ist omicron_{grund}_{collapsed?}.csv.
- Detaillierte Auswertung auf Bundeslandebene von Moritz Gerstung - leider nicht stets aktuell
The underlying files that I use as input are licensed by RKI under CC-BY 4.0, see more details here: https://github.com/robert-koch-institut/SARS-CoV-2-Sequenzdaten_aus_Deutschland#lizenz.
The software here is licensed under the "Unlicense". You can do with it whatever you want.